Die Erbinformation des Menschen besteht aus zirka 3 Milliarden Nukleotidbausteinen. Jeweils 1.000 bis 3.000 solcher Buchstaben bilden ein Gen. Die Begriffsdefinition des Gens ist nicht ganz einheitlich.
Gendefinition. In der klassischen Genetik bezeichnete man als Gen einen Erbfaktor, welcher ein Merkmal definiert. Heute wird ein Gen als chemisch definierter Abschnitt eines DNS-Stranges bezeichnet, der in eine Boten-RNS (Messenger-RNS, mRNS) übersetzt wird, die danach die Matrize für die Synthese eines spezifischen Eiweißes bildet. Diese kodierenden Teile des Gens werden als Exon bezeichnet. Alle kodierenden Abschnitte machen zusammen nur wenige Prozent des Genoms aus. Die nichtkodierenden „stummen“ Teile eines Gens, die Introns, bestehen aus den gleichen Bausteinen wie die der kodierenden Teile. Sie sind vielfach für die verschiedensten Regulationsaufgaben verantwortlich. Wir werden im Zusammenhang mit der Regelung der Genaktivität noch mehrmals im Detail auf diese Problematik zurückkommen, weil sie für das Verständnis der molekularen Vielfalt und Individualität von zentraler Bedeutung ist.
Betrachten wir nun den Weg der kodierenden genetischen Information der DNS eines Gens, wie sie im Kern bzw. den Mitochondrien vorliegt, bis zum fertigen Eiweißmolekül (Polypeptid), ohne vorerst die vielfältigen Regulationsmöglichkeiten in Betracht zu ziehen. Der Weg der genetischen Information von der DNS zum fertigen Protein wird als das „zentrale Dogma der Molekularbiologie“ bezeichnet. Der prinzipielle Ablauf ist in Abb. 7 und Abb. 8 skizziert.
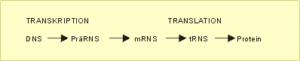
Vorweg sei bemerkt, daß die genetischen Informationen für das herzustellende Protein, die Exons, nicht in ununterbrochener Reihenfolge hintereinander vorliegen. Sie werden wiederholt durch Bereiche ohne entsprechende Informationen für die Synthese spezifischer Eiweiße, nämlich die erwähnten Introns, getrennt. Letztere werden zu einem späteren Zeitpunkt herausgetrennt.
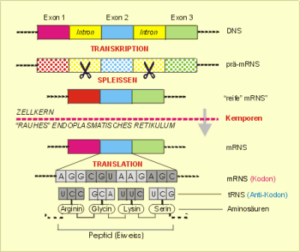
Transkription im Zellkern. In einem ersten Schritt, der Transkription, wird die DNS in eine vorläufige instabile Messenger-RNS (prä-mRNS) umkopiert, die noch alle Exons und Introns enthält. Der daran anschließende Prozeß des Spleißens – nämlich das Herausschneiden der nichtkodierenden Introns – stellt bereits auf dieser frühen Stufe der molekularen Informationsverarbeitung eine wichtige Quelle molekularer Differenzierung dar, da unter bestimmten Bedingungen die Exons nach dem Spleißen nicht wieder in der ursprünglichen Reihenfolge zusammengesetzt oder mit Exons aus anderen Genen kombiniert werden. Wir werden dies bei der Besprechung der Synthese spezifischer Antikörper im Zusammenhang mit dem alternativen Spleißen noch näher kennenlernen. Erst nachdem die Introns enzymatisch herausgeschnitten (gespleißt) wurden, haben wir die reife mRNS vor uns, die durch die Kernporen aus dem Kern auswandert und die endgültige Matrize für die Eiweißsynthese im endoplasmatischen Retikulum in der Nähe – aber außerhalb – des Zellkerns bildet.
Die Transkription wird durch durch das Enzym RNS-Polymerase bewerkstelligt. Für die Aufklärung der molekularen Zusammenhänge bei der Transkription erhielt im Herbst 2006 der Chemiker Roger D. Kornberg von der Universität Stanford (USA) den Nobelpreis für Chemie.
Translation außerhalb des Zellkerns. Translation bedeutet in diesem Zusammenhang: Übersetzung der Informationen, die in den Nukleotidsequenzen der Boten-RNS stecken, in Ketten von Aminosäure-Bausteinen, der Basis der Eiweißstrukturen. Dazu muß man wissen, daß die einzelnen Aminosäuren zum Transport an kleine Transfer-RNS-(tRNS)-Moleküle gebunden sind, die jeweils drei Nukleotide (Tripletts) tragen, die die gebundene Aminosäure charakterisieren und das Antikodon bilden. Durch enzymatische Prozesse werden die tRNS-Triplett-Aminosäurekomplexe komplementär an die Tripletts der mRNS-Matritze (Kodons)gebunden und miteinander verknüpft (Tab. 1). Auf diese Weise entsteht – entsprechend dem genetischen Kode – die Primärstruktur eines Eiweißmoleküls.
| Aminosäure | Abkürzung | Kode-1 | Kode-2 | Kode-3 | Kode-4 |
|---|---|---|---|---|---|
| Alanin | Ala (A) | GCU | GCC | GCA | GCG |
| Arginin | Arg (R) | AGG | AGA | ||
| Asparagin | Asn (N) | AAC | AAU | ||
| Asparaginsäure | Asp (D) | GAU | GAC | ||
| Cystein | Cys (C) | UGU | UGC | ||
| Glutamin | Gln (Q) | CAA | CAG | ||
| Glutaminsäure | Glu (E) | GAA | GAG | ||
| Glycin | Gly (G) | GGU | GGC | GGA | GGG |
| Histidin | His (H) | CAC | CAU | ||
| Isoleucin | Ile (I) | AUA | AUC | AUU | |
| Leucin | Leu (L) | UUA | UUG | ||
| Lysin | Lys (K) | AAG | AAA | ||
| Methionin | Met (M) | AUG | |||
| Phenylalanin | Phe (F) | UUU | UUC | ||
| Prolin | Pro (P) | CCU | CCA | CCC | CCG |
| Serin | Ser (S) | AGC | AGU | ||
| Threonin | Thr (T) | ACG | ACA | ACC | ACU |
| Tryptophan | Trp (W) | UGG | UGC | ||
| Tyrosin | Tyr (Y) | UAU | UAC | ||
| Valin | Val (V) | GUA | GUC | GUU | GUG |
| STARTCODON | AUG | ||||
| STOPCODON | UAA | UAG | UGA |
Dem Körper stehen 20 verschiedene Aminosäuren zur Verfügung, aus denen alle möglichen Aminosäureketten (kurze Peptide als auch Eiweiße) synthetisiert werden können. Da die 43 Kombinationen der 4 Basen in den Tripletts 64 Möglichkeiten ergeben aber nur 20 verschiedene Tripletts benötigt werden, um alle Aminosäuren zu kodieren, ist das System gewissermaßen überdimensioniert. So können die einzelnen Aminosäuren nicht nur durch ein einziges Triplett kodiert sein, sondern durch verschiedene Trinukleotide (Tab.1). Daneben werden für die Kodierung des Anfangs und des Endes der Peptidkette Start- bzw. Stopkodons verwendet.
Bei der Entwicklung bestimmter Antibiotika macht man sich die Erkenntnisse über den Ablauf der Kodierung der Eiweißsynthese zunutze, indem chemische Abkömmlinge (Analoga) einer Aminosäure dazu benutzt werden, den Prozess der Peptidsynthese in Bakterien zu unterbrechen. Diese sogenannten Analoga sind so synthetisiert, dass sie z.B. während der Peptidsynthese mit dem Vorgänger in der Peptidsequenz eine Verbindung eingehen können, mit dem Nachfolger jedoch nicht. Die pharmakologische Wirkung besteht also darin, dass im krankmachenden Bakterium die normale Synthese des Polypeptids bzw. Eiweißes abgebrochen wird, was zum Absterben des Bakteriums führt.
Die oben beschriebenen Einzelschritte umfassen nur wesentliche Gesichtspunkte zur Beziehung zwischen Nukleotidsequenz und Eiweißsynthese. An der Erforschung dieses Themengebietes waren und sind Tausende von Wissenschaftlern beteiligt. Neuerdings ist auch die Industrie in starkem Maße mit Milliardeninvestitionen an der Forschung als auch an der Produktion von Pharmaka, Bioprodukten (grüne, rote, weiße und graue Gentechnik) an dieser Entwicklung beteiligt. Soviel ist heute bereits sicher: Die molekularbiologische Wissenschaft und Technik wird unser ferneres Leben in weitaus stärkerem Maße beeinflussen als die sich heute rasant entwickelnde Informationstechnik, vor allem deshalb, weil sie unmittelbar – in positiver wie negativer Hinsicht – in unser physisches Leben eingreifen wird.
Fazit: Transkrption: Die in der DNS gespeicherte genetische Information wird in Form einer Kopie, der Boten-RNS (Messenger-RNS, mRNS) aus dem Zellkern in das sog. rauhe endoplasmatische Retikulum, den Ort der Eiweißsynthese, transportiert. Translation: Mit Hilfe kurzer Tranfer-RNS-Moleküle werden die einzelnen Aminosäuren entsprechend der Nukleotidsequenz der mRNS-Matrize zu einer Peptidkette, der Primärstruktur der Eiweiße, vereinigt. Der beschriebene Vorgang wird als das „zentrale Dogma der Molekularbiologie“ bezeichnet und ist für alle Lebewesen gültig.
