Allele. Als Allele bezeichnet man molekulare Varianten eines kodierenden Gens bzw. Genortes. Sie entstehen, wenn ein oder mehrere Nukleotidpaare gegen andere ausgetauscht werden (z.B. A-T gegen G-T) oder verloren gehen (vgl. auch Tab. 11). Die Gesamtheit aller Allele wird als Allelie bezeichnet und bestimmt den Umfang des molekularen Polymorphismus – die molekulare Vielgestaltigkeit eines Individuums. Je stärker dieser Polymorphismus ausgeprägt ist, desto besser ist eine Population in der Lage, sich ihrem Lebensraum anzupassen. In großen kollektiven Datenbanken wie der „Allele Frequency Database (ALFRED)“ werden diese Informationen weltweit gesammelt und der Öffentlichkeit für wissenschaftliche Zwecke zur Verfügung gestellt.
| Test-Antiserum | |||
|---|---|---|---|
| Testzelle | anti-A | anti-B | anti-A+anti-B |
| Blutgruppe A | POS | neg | POS |
| Blutgruppe B | neg | POS | POS |
| Blutgruppe AB | POS | POS | POS |
| Blutgruppe 0 | neg | neg | neg |
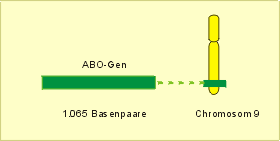
Blutgruppen. Zu den leichtverständlichen Beispielen, um den Begriff Allel zu verdeutlichen, zählen die Allele des AB0-Blutgruppensystems. Auf Chromosom 9 des Menschen existiert eine Nukleotidsequenz, die die Synthese der Merkmale der AB0-Blutgruppen kodiert (Abb. 19). Letztere sind biochemisch gesehen Glykoproteine oder Glykolipide an der Oberfläche der Zellmembran von roten Blutkörperchen (Erythrozyten), die durch spezifische Antikörper (anti-A oder/und anti-B) verklumpt werden können (Agglutinationsreaktion). Auf diesem Prinzip beruht die Blutgruppenbestimmung (Tab. 7).
| Blutgruppen | ||||
|---|---|---|---|---|
| O | A | B | AB | |
| Deutsche | 41 | 43 | 11 | 5 |
| Peruanische Indianer | 100 | 0 | 0 | 0 |
| Mayas | 98 | 1 | 1 | 1 |
| Blackfoot (nordam. Indianer) | 17 | 82 | 0 | 1 |
| Buriaten | 33 | 21 | 38 | 8 |
| Kalmücken | 26 | 23 | 41 | 11 |
| Tschuwaschen | 30 | 29 | 33 | 7 |
Hinsichtlich der Verteilung der AB0-Blutgruppen auf der Erde bestehen von Region zu Region zum Teil beträchtliche Unterschiede (Tab. 8). In der ersten Zeile ist die Verteilung der AB0-Blutgruppen in einer deutschen Stichprobe dargestellt. Ein ganz anderes Bild erhält man, wenn man bestimmte eingeborene Populationen Südamerikas betrachtet. In diesen Populationen findet man z.B. fast ausschließlich Individuen mit der Blutgruppe 0. In einem anderen nordamerikanischen Indianerstamm sind nur die Blutgruppen 0 und A vertreten. In Zentralasien finden wir Populationen mit einer hohen Frequenz von Individuen mit der Blutgruppe B.
| Phänotyp (Blutgruppe) | Genotyp |
|---|---|
| A | AA oder A0 |
| B | BB oder B0 |
| AB | nur AB |
| 0 | nur 00 |
Jeder Mensch erbt jeweils von einem seiner beiden Elternteile eines der beiden Allele – entweder A oder B, beide (AB) oder keines von beiden (0). Sind diese Allele von Vater und Mutter identisch (AA oder BB), bezeichnet man das Individuum als homozygot für dieses Allel, sind sie unterschiedlich als heterozygot für dieses Allel. Die ererbte Genkombination bildet den Genotyp der Blutgruppe. Da jedes der beiden Merkmale dominant vererbt wird, sich also durchsetzt und das Merkmal 0 stumm ist, kommt es je nach Kombination der väterlichen und mütterlichen Keimzellen zu einer neuen Kombination der Merkmale, den Phänotyp, der bei der Blutgruppenbestimmung erfaßt wird. Den Zusammenhang zwischen Phänotyp und Genotyp erläutert Tab. 9.
| Kind | ||||
|---|---|---|---|---|
| Eltern | A | B | AB | 0 |
| A x A | 94% | ~ | ~ | 6% |
| A x B | 19% | 19% | 56% | 6% |
| A x AB | 50% | 13% | 37% | ~ |
| A x 0 | 75% | ~ | ~ | 25% |
| B x B | ~ | 94% | ~ | 6% |
| B x AB | 13% | 50% | 38% | ~ |
| B x 0 | ~ | 75% | ~ | 25% |
| AB x AB | 25% | 25% | 50% | ~ |
| AB x 0 | 50% | 50% | ~ | ~ |
| 0 x 0 | ~ | ~ | ~ | 100% |
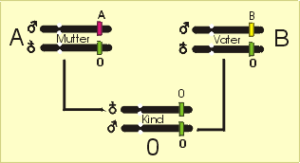
Eine lehrreiche (und gefürchtete) Prüfungsfrage ist: Kann ein Kind mit der Blutgruppe 0 einen Vater mit der Blutgruppe B haben. Antwort: ja. Die richtige Lösung ergibt sich daraus, daß der Mensch von jedem Chromosom ein Paar besitzt, wovon ein Chromosom wiederum von den Keimzellen des Vaters und eines von der Mutter stammt. Abb. 20 verdeutlicht diesen Zusammenhang: Hat der Vater z.B. den Genotyp B0 und die Mutter den Genotyp A0 oder B0 oder 00 und das Kind erbt von beiden Eltern das Allel 0 (nicht A, nicht B), so führt das beim Kind zu Blutgruppe 0 als Phänotyp. Also ganz einfach (vgl. auch Tab. 9a).
| AB0-Blutgruppen | Lutheran-Blutgruppen |
| Cartwright-Blutgruppen | MNSs-Blutgruppen |
| Colton-Blutgruppen | P-Blutgruppen |
| Diego-Blutgruppen | Rhesus-Blutgruppen |
| Scianna-Blutgruppen | Dombrock-Blutgruppen |
| Gerbich-Blutgruppen | Vel-Blutgruppen |
Übrigens gibt es neben dem AB0-System weitere Blutgruppensysteme mit mehr als 200 Blutgruppen-Merkmalen, die auf den roten Blutkörperchen vorkommen können (Tab. 10), deren Allele ebenfalls als Kombination mütterlicher und väterlicher Antigene an die Nachkommen weitergegeben werden und dort individuelle molekulare Muster bilden. Für die Blutgruppenübereinstimmung sind in der täglichen Transfusionspraxis jedoch nur wenige Blutgruppenmerkmale (hauptsächlich ABO und Rh) von vordergründiger praktischer Bedeutung. Allerdings können unter bestimmten Umständen Inkompatibilitäten (Nichtübereinstimmungen) hinsichtlich der Blutgruppe zwischen Blutspender- und Empfänger für solche Nicht-AB0-Merkmale zu lebensdrohlichen Transfusionszwischenfällen führen. Soweit also die „einfachen“ Verhältnisse bei roten Blutzellen.
Gewebeantigene. Ähnlich den roten Blutkörperchen besitzen alle Zellen an ihrer Oberfläche weitere allele Strukturen, die sich von Individuum zu Individuum unterscheiden können. Besonders gut untersucht sind die sog. Gewebeverträglichkeitsantigene (Histokompatibilitätsantigene oder Transplantationsantigene), so genannt wegen ihrer Bedeutung für die Organtransplantation. Wir finden sie auf fast allen Körperzellen mit Ausnahme der roten Blutkörperchen. (Wir werden auch hier wieder für die Bezeichnung von Zelloberflächenmolekülen den Begriff Antigen benutzen.)
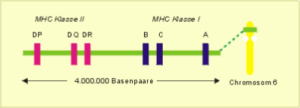
Hier nur so viel: Auf dem Chromosom 6 des Menschen befinden sich mehrere Genorte, die die Synthese der Transplantationsantigene kodieren (Abb. 21). Zusammen bilden sie den sog. Haupthistokompatibilitätskomplex (MHC), so genannt wegen seiner Bedeutung für die Abschätzung der Gewebeverträglichkeit (Histokompatibilität) im Rahmen der Organtransplantation.
| HLA-X*01 | ~~~ ACGTGGTCATAATGCTTCCAGACA ~~~ |
| HLA-X*02 | ~~~ ––––C––––––––––––––––––– ~~~ |
| HLA-X*03 | ~~~ –––––––A–––––––––––––––– ~~~ |
| HLA-X*04 | ~~~ –––––––––––––––––TT––––– ~~~ |
| HLA-X*05 | ~~~ ––––C––A–––––––––––––––– ~~~ |
| HLA-X*06 | ~~~ ––––C––––––––––––TT––––– ~~~ |
| HLA-X*07 | ~~~ –––––––A–––––––––TT––––– ~~~ |
| HLA-X*08 | ~~~ ––––C––A–––––––––TT––––– ~~~ |
Für die einzelnen Genorte des MHC besteht ein ausgeprägter Polymorphismus. Im Gegensatz zu den roten Blutkörperchen, wo wir nur drei Allele für den Genort der AB0-Blutgruppen finden, können hier allein für einen der für die klinische Praxis bedeutsamen Genorte hunderte im Labor bestimmbare Allele existieren. Für die ganz Mutigen unter Ihnen kann hier die umfangreiche Liste aller bis heute bekannten der Allele (Klasse I und Klasse II; Stand 2014)) des MHC empfohlen werden, die im Internet veröffentlicht wurde. Schon sie allein bildet den überzeugenden Beweis für die möglichen individuellen molekularen Differenzen zwischen zwei Individuen. Der Unterschied liegt vielfach nur im Austausch eines Nukleotids in einem bestimmten Genbereich (Prinzip vgl. Tab. 11). Wenn man dazu noch berücksichtigt, daß die Allele der einzelnen Genorte immer in unterschiedlichen Kombitionen (Mustern) auftreten, so potenziert sich die molekulare Vielfalt um weitere Größenordnungen.

Bedenkt man schließlich, daß von den Eltern jeweils zwei verschiedene dieser Allel-Muster vererbt werden, so kommt man leicht auf mehrere Millionen möglicher Muster. Die praktische Folge: Es ist schwierig, geeignete Organspender für einen bedürftigen potentiellen Organempfänger zu finden, der in allen bestimmbaren Gewebemerkmalen, die sich bisher für die Transplantation als bedeutend erwiesen haben, mit den Gewebemuster des Empfängers übereinstimmt. (vgl. Abschnitt „Transplantation und Transfusion“ )
Wie kann man sich eine solche Allelbestimmung (Gewebetypisierung) vorstellen? Man unterscheidet zwei Verfahren: ein serologisches und ein molekularbiologisches. Das serologische Verfahren verwendet – wie bei der Blutgruppenbestimmung – Testseren mit Antikörpern definierter Spezifität, das zweite Verfahren verwendet sog. Primer, das sind relativ kurze DNS-Stücke mit definierter Nukleotidsequenz, die komplementär zu bestimmten kodierenden DNS-Sequenzen der einzelnen Loci der HLA-Antigene sind.
Das seit vielen Jaren etablierte serologische Verfahren weist Antigenstrukturen – also spezifische Glykoproteine – an der Oberfläche von Lymphozyten nach. Es benötigt für eine Untersuchung nur 1µl Untersuchungsmaterial, das mit 1 µl Testserum vermischt wird. Nach einer halben Stunde Inkubation werden für eine weitere halbe Stunde 5µl „Komplement“ (neutrales Kaninchenserum) zugefügt, das einen Eiweißkomplex enthält, der die Lymphozyten zerstört, falls sich die Testantikörper an die Zelle gebunden haben. Die Zerstörung der Zellmembranen als auch das Eindringen eines zugegebenen Farbstoffs in die Zellen zeigt unter dem Mikroskop eine positive Reaktion an. Mit einem Satz definierter Testseren kann auf diese Weise das HLA- Muster der Lymphozytenprobe bestimmt werden.
Das molekularbiologische Verfahren setzt ein wesentlich umfangreicheres Arsenal an Geräten voraus und weist spezifische Nukleotidsequenzen im Zellkern der untersuchten Zellen nach. Zuerst muß die DNS aus den Zellen der Probe gewonnen und mit den spezifischen Primern versetzt werden. In einem zyklischen Verfahren (PCR; vgl. Abschnitt „Nukleinsäuren“) erfolgt die Vervielfältigung der spezifischen DNS-Abschnitte. Die entstandenen Produkte werden dann in einem elektrophoretischen Verfahren aufgetrennt und die entstandenen Banden analysiert. Mit einem Satz definierter Primer kann das HLA- Muster der Lymphozytenprobe bestimmt werden, wobei unter Verwendung entsprechender Primer ein höherer Grad an Differenzierung der Allele für die einzelnen Genorte erreicht werden kann als beim serologischen Verfahren. Allerdings muß man sich dabei immer im Klaren darüber sein, daß der Nachweis der genetischen Information (Genotyp) bei der „hochauflösenden“ Analyse nicht zwangsläufig mit den an der Zelloberfläche exprimierten molekularen Strukturen (Phänotyp) identisch sein muß, da die genetische Informationen nicht in jedem Fall auch zu phänotypisch nachweisbaren Produkten führt.
Aus praktischen Gründen werden die Bestimmungen der Gewebe-Allel-Muster an Lymphozyten aus dem Blut oder der bei der Organentnahme gleichzeitig entnommenen Gewebeprobe aus der Milz vorgenommen, da sie wesentlich leichter und zerstörungsfrei zu gewinnen sind als entsprechendee Zellen aus den zu transplantierenden Organen. Die nachzuweisenden Strukturen an der Oberfläche der Lymphozyten werden nach ihren Trägern Human-Lymphozyten-Antigene (HLA-Antigene) genannt.
Bei der Verwendung von Lymphozyten für die HLA-Gewebetypisierung anstelle von Organzellen wird stillschweigend vorausgesetzt, daß die an Lymphozyten bestimmten Gewebeantigene repräsentativ für die Allelmuster in den entnommenen Organen (z.B. Niere oder Herz) sind. Dies ist in der Regel auch richtig aber nur die halbe Wahrheit. Erstens weicht die Dichte der HLA-Antigene in den Organgeweben – insbesondere in deren Blutkapillaren z.T. – sehr stark von derjenigen an der Oberfläche von Lymphozyten ab, und zweitens gibt es neben dem HLA-System noch andere wichtige Allel-Systeme, deren Merkmale nicht auf den Lymphozyten repräsentiert sind – also somit nicht bestimmt werden – aber nachweislich zur Fremderkennung nach einer Organtransplantation führen und folglich zur Transplantatabstoßung beitragen können. Dazu gehört z.B. ein Antigensystem, dessen Allele bevorzugt auf den Endothelzellen zu finden sind, die die Innenwand der Blutgefäße des Spenderorgans auskleiden und bei Alleldifferenzen zwischen Empfänger und Spender mit hoher Wahrscheinlichkeit zur immunmologischen Fremderkennung und zu entsprechenden Abwehrreaktionen führen können.
Enzymgruppen und Immunglobuline (Antikörper): Zum Abschluß dieses Abschnitts soll nicht unerwähnt bleiben, daß es Polymorphismus auch bei bestimmten Enzymen (Tab. 12) und den später zu behandelnen Antikörpern gibt (vgl. Abschnitt „Antigene und Antikörper„). Beiden Molekülgruppen ist gemeinsam, daß sie eine hohe funktionelle Spezifität aufweisen. Funktionell gleiche Moleküle können sich jedoch durch ihren Polymorphismus von Individuum zu Individuum in ihrer molekularen Struktur nachweislich unterscheiden.
| Adenosindesaminase | Adenylatkinase |
| Esterase D | Glutamat-Pyruvat-Transaminase |
| Glyoxalase I | Phosphoglukomutase |
| 6-Phosphogukonat-Dehydrogenase | Phosphoglykolatphosphatase |
| Phosphoglyzeratmutase | saure Erythrozytenphosphatase |
Das Human Genome Diversity Project. Neben dem weiter oben genannten bekannten Human Genome Project (HGP), das sich der Aufklärung der Nukleotidsequenz des menschlichen Genoms verschrieben hat, gibt es seit 1993 das (inzwischen eingestellte) sogenannte Human Genome Diversity Project(HGDP), das die Identifizierung und Allel-Verteilung von möglichst vielen verschiedenen Polymorphismen in möglichst vielen verschiedenen Populationen und Ländern der Erde zum Ziel hat. Beide Projekte haben organisatorisch nichts miteinander zu tun. Wie schon der Name des Projekts andeutet, geht es hierbei um die Erfassung der genetischen Unterschiede zwischen bestimmten Populationen, um eine Art genetischen Weltatlas zu erstellen. Dieser sollte dann die Verschiedenheit der Erbinformationen der gesamten Menschheit enthalten.
Die am HGDP-Projekt beteiligten Wissenschaftler und Organisationen versichern zwar, die genetischen Proben oder irgendein daraus entstandenes Produkt nicht patentieren zu lassen, weil man lediglich auf Wissen und nicht auf Profit aus sei. Die Erfahrung zeigt jedoch, daß bereits heute einzelne Gene und sogar ganze DNA-Sequenzen dank des Patentrechts im Besitz großer Konzerne sind.
Das Genographic Project. Das im Frühjahr 2005 begonnene Genographic Project der US-amerikanischen National Geographic Society an 100.000 Personen aller Kontinente nutzt die Verwendung mitochondrialer Marker und Marker auf dem Y-Chromosom, um aus den daraus abgeleiteten Verwandschaftsbeziehungen die historischen Wanderungsbewegungen der Menschheit zu kartieren. Diese weltweit angelegte Studie soll im Gegensatz zum Human Genome Diversity Project ihre Informationen der Öffentlichkeit zur Verfügung stellen.
Fazit: Allele sind Varianten eines Genorts. Beispiele sind die Blut-, Gewebe- und Enzymgruppen. Nimmt man die bereits heute bestimmbaren Allele zusammen, so wird offensichtlich, daß die Anzahl der theoretisch möglichen Allelmuster außerordentlich groß ist und in hohem Maße zur molekularen Vielfalt – insbesondere auf Eiweißebene – beiträgt. Auf dem Gebiet der Organtransplantation ist es deshalb schwer, passende Spender-Empfängerpaare zu finden.
