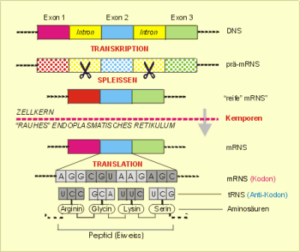
Bisher wurde deutlich, daß entsprechend den physiologischen Erfordernissen immer andere Muster von Genen an- und abgeschaltet werden. In Anlehnung an den Begriff Genom wird die Gesamtheit der zu einem bestimmten Zeitpunkt abgelesenen (transkribierten) Gene als Transkriptom bezeichnet, die Gesamtheit aller zu einem Zeitpunkt in einem Individuum daraus abgeleiteten Proteine als Proteom.
An dieser Stelle bietet sich die Gelegenheit, ein Verfahren kennenzulernen, mit dem es möglich ist, festzustellen, welche Gene zu einem bestimmten Zeitpunkt in einem bestimmten Zelltyp angeschaltet sind (transcript imaging, Abb. 15). Die Methode beruht auf der „Zurückschreibung“ der instabilen Boten-RNS des Zellpräparates in sogenannte Copy-DNS (cDNS) mit Hilfe eines Enzyms (Reverse Transkriptase), das aus einem RNS-Retrovirus gewonnen wird. Die cDNS stellt also die Kopie eines Abschnitts aus der Boten-RNA dar. Die resultierende stabile intronfreie cDNS kann dann mit spezifischen Sonden analysiert werden. Solche Sonden sind kurze Nukleotidketten mit bekannter Sequenz, die mit komplementären DNS-Abschnitten reagieren können.

Mittels der Microarraytechnik (DNA-Chips, Biochips, Genchips) können gleichzeitig Zehnausende winziger Tröpfchen solcher verschiedener Sonden mit bekannter Spezifität (Anti-Nukleotidsequenz) auf wenige Quadratzentimeter große Trägerplatten aufgebracht werden. Nach Zugabe der vorher mit Fluoreszenzfarbstoffen markierten cDNS-haltigen Probe „fischen“ sich die Sonden die komplementären cDNS-Moleküle aus dem cDNS-Gemisch. Dieser Vorgang wird Hybridisierung genannt (Abb. 16). Nach Anregung mit UV-Licht leuchten die winzigen hybridisierten Sonden-Spots auf und können mittels eines Mikroscanners analysiert und unter bestimmten Umständen sogar quantifiziert werden (Abb. 17). Da die Spezifität der Sonden bekannt ist, kann auf die „angeschalteten“ Gene rückgeschlossen werden. Abweichungen der mRNS-Muster von solchen gesunder Probanden können z.B. Hinweise auf spezifische Erkrankungen geben. Es handelt sich um ein universelles Verfahren, das sowohl in der Grundlagenforschung als auch in der klinischen Praxis Anwendung findet. Biochips gehören gegenwärtig zu den fortschrittlichsten Methoden der Analytik von Biomolekülen. Wo bisher Hunderte oder Tausende von Einzelversuchen im Reagenzglas notwendig waren, finden diese nun auf winzigen Glasplatten statt.
Ziel der Wissenschaftler ist es, die Regulation der einzelnen Gene aufzuklären, um die molekularen Netzwerke zu verstehen, die den komplexen Lebensprozessen zugrunde liegen. Zu diesem Zweck wurde in internationaler Zusammenarbeit, bereits 1996 innerhalb des Human Genom Projekts das Deutsche cDNA Konsortium gegründet.
Fazit: Die Ablesung der genetischen Information ist ein hochdynamischer Prozeß, der durch spezielle regulative Moleküle gesteuert wird. Die Gesamtheit aller zu einem Zeitpunkt abgelesenen („angeschalteten“) Gene wird als Transkriptom bezeichnet. Mit Hilfe eines speziellen Enzyms (Reverse Transkriptase) können aus der jeweiligen Boten-RNS DNS-Moleküle erzeugt werden. In einem Mikrohybridisierungsverfahren (Microarraytechnik) kann man diese Moleküle sichtbar machen.
